引言
矢量化编程是提高机器学习算法速度的一种有效方法。例如,在线性回归模型中,我们已经求得了回归参数$\theta$和$b$,现在我们需要用该模型对一批数据进行预测,一个朴素的方法是利用for循环实现:
y = [theta * x + b for x in X]
但实际上,numpy和pytorch等框架提供了矩阵乘法的接口,避免了显示的for循环,不但可以增加代码可读性,还可以提高代码运行效率:
y = X @ theta + b
除了上面的线性回归,逻辑回归、支持向量机、神经网络模型,都是可以通过这种方法去加速,因为它们的判别函数都是矩阵乘法的衍生物。与此相反,kNN算法由于需要对每一个样本求最近邻样本,决策树的每个样本需要在每个节点进行判断,这样的逻辑判别的判别函数是难以用过矩阵运算去加速的,我们这里主要想讨论决策树在预测时的加速方法。
避免循环
假设我们的决策树分类器对鸢尾花数据集进行划分,形成了下面的决策树:
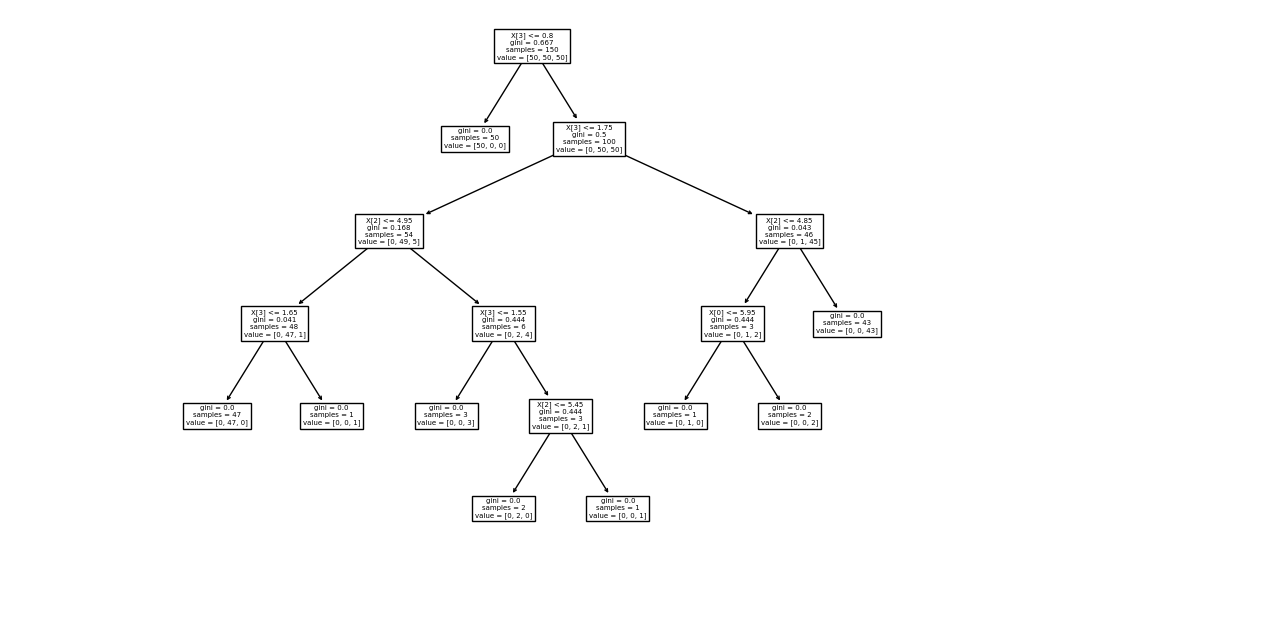
现在对新的100个数据,我们想对其进行预测。我们似乎只能用for循环,将每个样本送入树中,判断其最后落入的叶节点属于哪个类。我们这样定义树节点:
class TreeNode:
def __init__(self, cls=None, depth=0) -> None:
# 该节点的划分准则,这里是特征的序数
self.split_attr: int = -1
# 该节点的划分阈值,不大于该值的数据落入左子节点,否则落入右子节点
self.shreshold: float = None
# 连续属性下,只有左右两个子节点
self.child = (None, None)
# 叶节点的类别,如果样本落入该叶节点,则判断其为cls类
self.cls: int = cls
# 节点深度,用于预剪枝
self.depth = depth
对于一个输入数据,我们通过循环(或递归)的方式来让其在树结构移动:
def predict_one(root, x):
'''
Parameter
---------
root: 决策树根节点
x: 输入数据
'''
node = root
while node.cls == None
# 我们在生成树的时候,只有叶节点的cls才不为None
left = x[node.split_attr] <= node.shreshold
if left is True:
node = node.child[0] # 左子节点
else:
node = node.child[1]
return node.cls
从而对于一大批输入数据,我们只能依次判断:
def predict(root, X):
pred = [predict_one(root, x) for x in X]
return pred
这样太死板而且太慢,我们想避免for循环,但通过矩阵优化的路被堵死了,我们只能从并行计算上去想办法,一种是进程上的并行:在sklearn的随机森林等模型中,joblib.parallel模块被用于for循环的优化。
下面是优化的示例:
from joblib.parallel import Parallel, delayed
import numpy as np
def matrix_func(n):
A = np.random.randn(n, 100)
B = np.random.randn(100, 100)
C = np.random.randn(100, n)
D = A @ B @ C
E = D @ D @ D
return E
# 对l = [matrix_func(i) for i in range(1, 10000)]的优化
l = Parallel(n_jobs=6)(delayed(matrix_func)(i) for i in range(1, 10000))
这一方法在大数据下效果很好,但随着数据规模减小,joblib在进程创建上的开销比重会增加,反而会导致计算速度的减小。
另一种是从算法实现上的优化,“并行”可以理解为多个样本同时送入决策树,类似决策树生成的过程,只不过这次不需要创建新的节点;样本最后会分布在各个叶节点,最后只要在每个叶节点收集测试样本即可获得预测结果。我们做一个不那么严谨的分析:假设我们的决策树深度为$d$,对于$N$个样本的输入,如果采用笨拙的单样本输入,时间复杂度是$O(Nd)$,而如果我们采用新算法,最坏情况就是决策树的每一个叶节点都有输入样本,因此时间复杂度为$O(2^d)$,也就是说新算法的时间只与树结构有关,当大量数据输入时,这一算法显然更佳。
算法实现
我们在原来实现的算法上进行修改:
-
TreeNode类中加上成员predict_list,用来记录落在某叶节点上的样本(在这里用对应的index代替,以节省空间); -
维护一个列表
leaf_list,用来存储生成决策树的叶节点; -
对
predict函数进行修改:class DecisionTreeClassifier: def predict(self, X): # 模拟上述过程,数据落到各个叶节点 DecisionTreeClassifier.recursion_split( self.root, X, np.arange(len(X)) ) pred = np.empty(len(X)) for leaf in self.leaf_list: if leaf.predict_list != None: pred[leaf.predict_list] = leaf.cls # 必须将predict_list清空,防止影响下一次预测 leaf.predict_list = None return pred @staticmethod def recursion_split(node, X, index_list): # 样本送入决策树后,递归划分样本直到到达叶节点 if node.cls != None: node.predict_list = index_list else: # 该分到node的左子节点的样本index index_left = X[:, node.split_attr] <= node.shreshold # 该分到右子节点的样本不需要重新计算,not一下就行 index_right = np.logical_not(index_left) DecisionTreeClassifier.recusion_split( node.child[0], X[index_left], index_left, ) DecisionTreeClassifier.recusion_split( node.child[1], X[index_right], index_right, )
效果验证
为了效果验证,我们保留了原来的predict方法,将新算法函数定义为predict_rec;用sklearn中的乳腺癌数据集作为训练数据:
from sklearn.datasets import load_breast_cancer
X, y = load_breast_cancer(return_X_y=True)
model = DecisionTreeClassifier()
model.fit(X, y) # 由于准确率不是终点,因此不考虑训练集和测试集的划分
然后多轮对数据进行预测,对累积时间绘图:
import matplotlib.pyplot as plt
from time import time
# 轮次
ITERATION = 5000
# 记录时间的列表
rec_list, iter_list = [0], [0]
t = time()
for i in range(EPOCHES):
model.predict_rec(X)
rec_list.append(time() - t)
for i in range(EPOCHES):
model.predict(X)
iter_list.append(time() - t)
plt.plot(rec_list, label="recursion")
plt.plot(iter_list, label="iteration")
plt.legend()
plt.show()
绘制出图像:
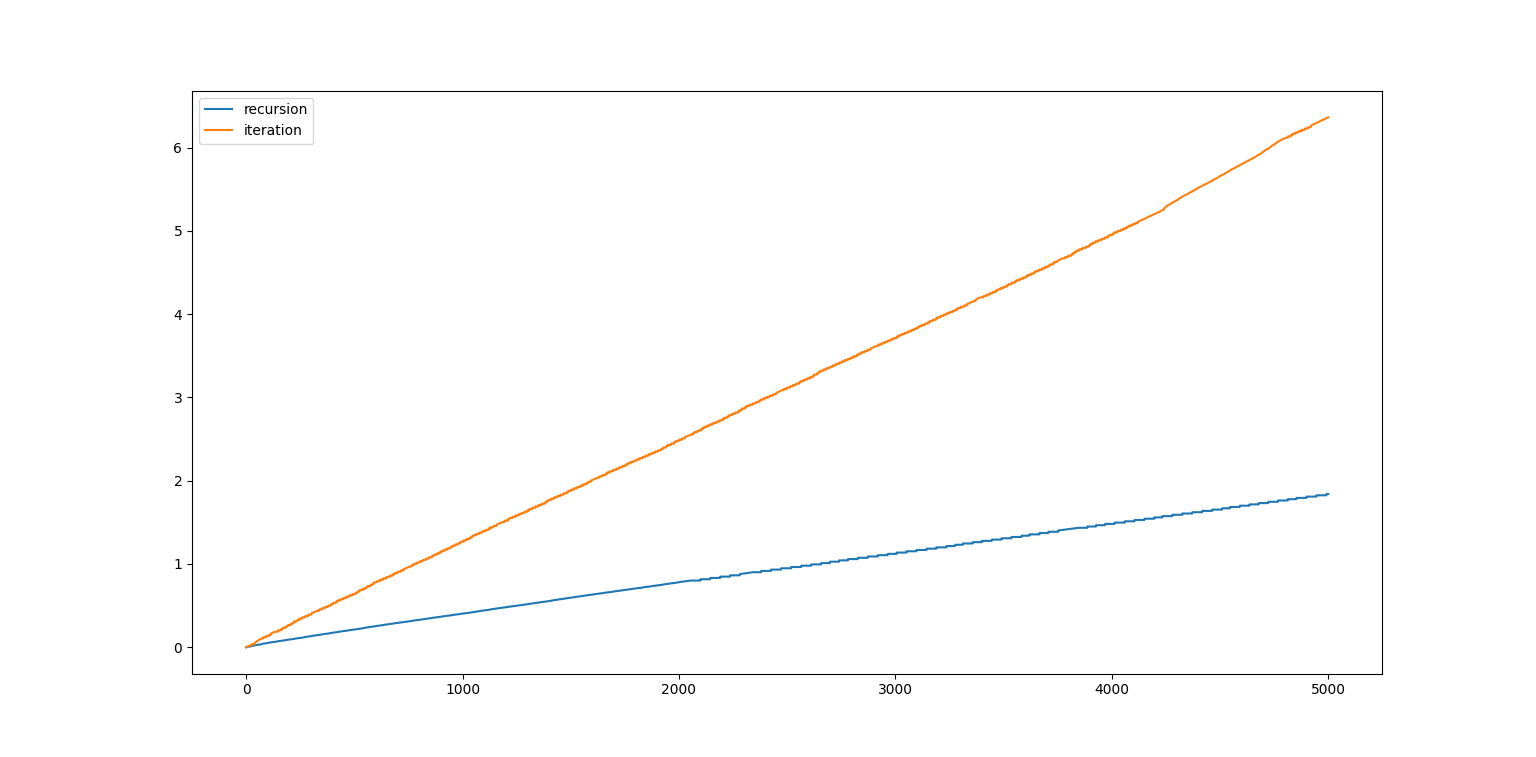
从斜率可以看出,我们的新算法将预测速度优化成原来的3-4倍,说明方法是有效的。
思考
注意到我们的新算法在实现时是一个尾递归,我们考虑将递归改为等价的循环,看是否可以进一步优化。可以发现这是一个深度优先搜索,所以需要用后进先出栈来辅助实现循环。恰好Python3的内置list通过提供append和pop方法,从而可以被我们作为栈使用:
def predict_dfs(self, X):
stack = []
# 根节点入栈
stack.append([self.root, X, np.arange(len(X))])
while len(stack) > 0:
node, data, id_list = stack.pop()
if node.cls != None:
node.predict_list = id_list
else:
# 该分到node的左子节点的样本index
index_left = X[:, node.split_attr] <= node.shreshold
# 该分到右子节点的样本不需要重新计算,not一下就行
index_right = np.logical_not(index_left)
# 子节点入栈
stack.append([
node.child[0],
data[index_left],
id_list[index_left],
])
stack.append([
node.child[1],
data[index_right],
id_list[index_right],
])
pred = np.empty(len(X))
for leaf in self.leaf_list:
if leaf.predict_list != None:
pred[leaf.predict_list] = leaf.cls
leaf.predict_list = None
return pred
我们可以将三种算法(数据循环输入,递归树遍历,循环树遍历)进行比较,绘制类似上面的轮次-时间图像:
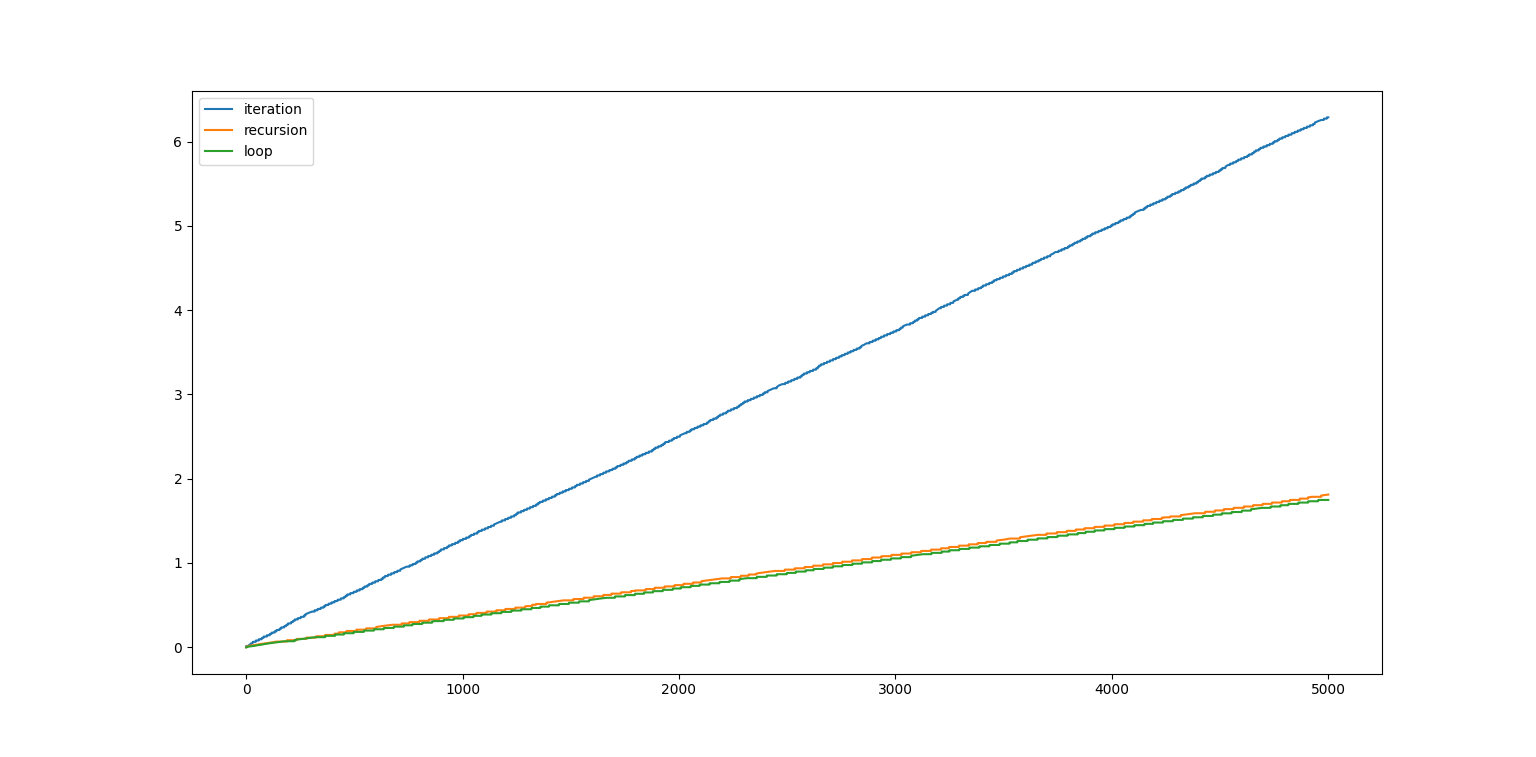
发现我们的循环优化并没有带来性能上的提升,笔者的一个想法是Python对递归进行了优化,导致和循环遍历的性能相近。